コアファシリティの利用について
共用機器の利用方法
① iSAL拠出機器(学内外研究者が利用可能):iSAL/KUMaCoから予約
10x Genomics社製 Chromium Controller
10x Genomics社製 Chromium X ハイスループットシステム
Bio-Rad社製 QX200 Droplet Digital PCRシステム
Takara Bio USA社製 SMARTer ICELL8 cx Single-Cell System
米国Sage Science社製 PippinHT
ダイアジェノード社製 MegaruptorR3
英国オックスフォード・ナノポアテクノロジーズ社製 PromethION24
Bio-Rad社製 C1000 Touch Thermal Cycler(課金なし)
京都大学医学研究支援センターのKUMaCoよりご予約ください。
※初めて利用される方は、KUMaCoで利用登録を行ってください。
(KUMaCo登録は毎年度更新必要)
② ASHBiグループのメンバー限定共通機器: 専用サイトから予約
利用登録には、予約システムのアカウント申請が必要です。アカウント申請は、所属グループのPIをCCに入れ、下記アドレスまでMailにてご連絡ください。
(宛先: ashbi-signac-office【@】mail2.adm.kyoto-u.ac.jp)
※アットマークのカッコを外し、半角英数に直してご使用下さい。
初めて使用する機器については、使用方法の説明や必要なトレーニングの案内を行います。
- 機器予約サイト(京都大学 学内限定)
料金表
利用時間
SignAC実験室機器利用可能時間:平日 9:00~17:00
解析サービスの利用方法
ASHBi SignACでは以下の解析サービスを提供しています。学内外の研究者が利用可能です。
- イルミナNGS解析
- RNA-seqライブラリー作製とNGS解析
- 英国オックスフォード・ナノポアテクノロジーズ社製 PromethION24
- 米国Pacific Biosciences社製 Sequel Ⅱe
- 米国アジレント・テクノロジーズ・インク製 Femto Pulse System
- 米国アジレント・テクノロジー社製 Agilent 4200 Tape Station
-Iso-Seqライブラリーの調整とシーケンス解析
-シーケンス解析のみ
-超微量RNAのクオリティーチェック
-サーモフィッシャー社Qubitを用いたNGSライブラリの定量とクオリティーチェック
申込方法
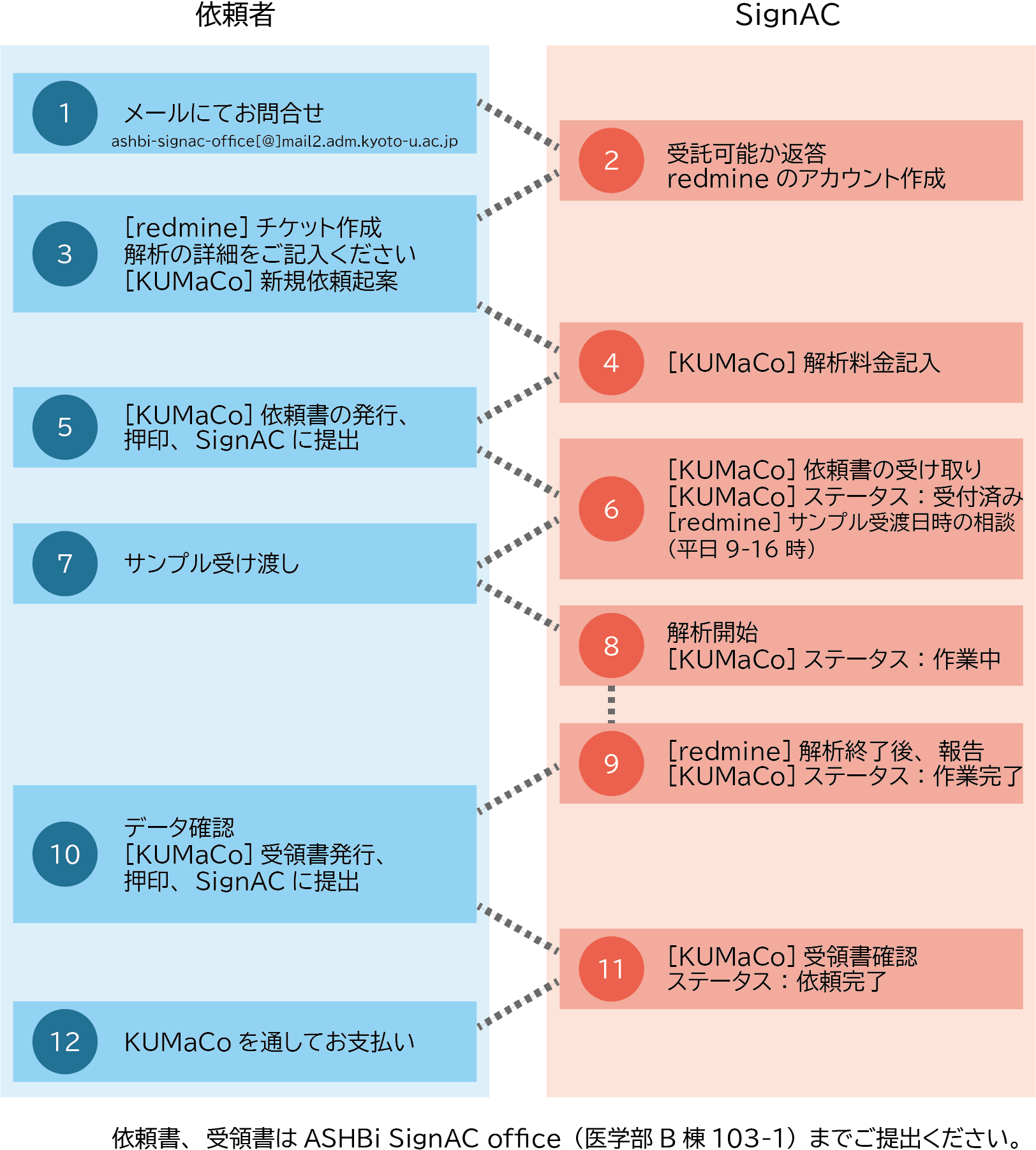
1. はじめてのご依頼は、あらかじめメールでご相談ください。 その際、RNA-Seq(受託)、イルミナNGS解析(受託)、その他相談のいずれになるのかを明記くださいませ。 (宛先: ashbi-signac-office【@】mail2.adm.kyoto-u.ac.jp) ※アットマークのカッコを外し、半角英数に直してご使用下さい。
2. 受託解析が可能な場合は、解析サービス管理システム(SignAC Redmine)のアカウントを発行いたします。 本システムから解析依頼の詳細項目についてご記載をお願いいたします。
3. 同時に、正式な解析依頼を、京都大学医学研究支援センターのiSAL/KUMaCoよりご依頼ください。
- KUMaCo(初めて利用される方は、利用登録を行ってください。登録は毎年度必要です。)
※SignAC Redmineをすでにご利用いただいている方については、上記のRedmineとKUMaCoからの依頼を同時に進めてください。
4. 依頼および解析内容の確定後に、KUMaCoより依頼書を印刷し、調製したサンプルとともに下記までお持ちください。
サンプル提出場所:医学部B棟1階101室・ASHBiゲノム情報解析コア
受付時間:平日 9:00~15:30
<受託解析の流れ>
イルミナNGS解析
NovaSeq6000、NextSeq550、iSeq100の各システムを利用したNGS解析を実施します。
調整済みライブラリーをご提出いただき、シーケンス結果を納品します。
インストール濃度はご指定ください。
以下の表は、llumina NovaSeq6000 Denature and Dilute Libraries Guide (1000000106351 v03)からの転載です。
NovaSeq6000のサンプル濃度については、こちらをご参照ください。
NextSeq550のローディング濃度は1.5-1.8pMを目安とすることが多いです。
iSeqについては、イルミナ社サイトをご参照ください。
NovaSeq6000参考インストール濃度
NovaSeq6000 NGSサンプル濃度 Standard workflow
| ライブラリーのタイプ | インストール濃度 (pM) | 提出サンプル濃度 (nM) |
| PhiX | 250 | 1.25 |
| Illumina DNA PCR-free library pool | 400–600 | 2–3 |
| TruSeq DNA PCR-free library pool | 175–350 | 0.875–1.75 |
| DNA PCR-amplified library pool | 300–600 | 1.5–3.0 |
| Single Cell | 250–500 | 1.25–2.5 |
NovaSeq6000 NGSサンプル濃度 XP (S4 レーンシーケンスの時のみ)
| ライブラリーのタイプ | インストール濃度(pM) | 提出サンプル濃度 (nM) |
| DNA PCRfree library pool | 115-235 | 0.575–1.175 |
| DNA PCRamplified library pool | 200-400 | 1.0–2.0 |
| Single Cell | 175-275 | 0.875–1.375 |
必要サンプル量
NovaSeq6000
すべてのサンプルをプールし、インストール濃度の5倍の濃度に調整し、以下に記載する指定量以上を提出。
SP/S1: 250μL以上
S2: 450μL以上
S4: 700μL以上
S4レーンシーケンス: 70μL以上
NextSeq550
4nM: 15μL以上
2nM: 30μL以上
1nM: 60μL以上
0.5nM: 120μL以上
すべてのサンプルをプールし、いずれかの濃度に希釈して提出
iSeq100
1nM: 15μL以上
データ受け渡しについて
ASHBi外の研究者にはデータ(通常fastqファイルとしますが、生データでも可)をHDDまたはフラッシュメモリーにコピーしてお渡しします。
fastqファイル納品の方はインデック情報の記載されたサンプルシートの提出をお願いします。
●Illumina Experiment Manager
>ダウンロードリンク >ユーザーガイドリンク ●以下のリンクからサンプルシート(見本)をダウンロードできますので、ご自由にご使用ください。
>サンプルシート(見本)のダウンロード >サンプルシート(記載例) index 2の向きにご注意ください。
参考→>リンク
RNA-seqライブラリー作製とNGS解析
- 原則100ngのトータルRNAから以下のキットを使用してライブラリー調製いたします。
NEBNext Poly(A) mRNA Magnetic Isolation Module[E7490]
NEBNext Ultra II Directional RNA Library Prep Kit [E7760]
NEBNext Multiplex Oligos for Illumina (96 Unique Dual Index Primer Pairs) [E6440] - ライブラリー作製には、Biomek i7自動分注ワークステーションを利用します。
- シーケンスは1サンプルあたりペアエンド50bp, 1500万リード以上を目安としております。
- fastqファイルを納品します。アダプタートリミングは行っていません。
持ち込みトータルRNAの要件
ボリューム:最低8μL
総量:最低300ng
濃度:10-150ng/μL、推奨50-100ng/μL
- RNA精製過程でDNase 処理することをお勧めしております。(必須ではありません)
- RNAの濃度は各自測定してください。(Qubit、NanoDrop等)
- SignACではライブラリー調整前にRNA integrityの測定は実施しません。なお、使用するライブラリー調整キットではRIN値7以上であることが推奨されています。各自Agilent Bioanalyzer等でこれを確認することを推奨します。(RNAの不具合で生じた問題は各自の自己責任とさせていただきます)
- Redmineにサンプル名(チューブに書いてある情報)、濃度を記入したエクセル等のファイルを提出してください。
- サンプル名には通し番号をつけ、チューブにはその番号も記載してください。
- 原則、各自記載の濃度をもとにライブラリー調整に進みます。
英国オックスフォード・ナノポアテクノロジーズ社製 PromethION24
>詳細
米国Pacific Biosciences社製 Sequel Ⅱe
>詳細
TapeStation/FemtoPulse による核酸の品質管理 (QC)
>詳細
ヒト指針対象研究について
- 研究計画書にSignACへの解析委託についてご記載ください。
- SignAC Redmine上で、研究計画書の承認番号を記入し、またSignACの遵守すべきことを記載してください。(記載いただいた内容がSignACでは対応不可能と判断される場合には解析を受付できないことをご了承ください)
解析一覧(料金表)
免責事項
成果発表について
SignACを利用しての成果発表時には謝辞の記載をお願いいたします。
(English)
We thank Single-cell Genome Information Analysis Core (SignAC) at WPI-ASHBi, Kyoto University, for their supports.
(Japanese)
京都大学高等研究院ヒト生物学高等研究拠点 (WPI-ASHBi),単一細胞ゲノム情報解析コア (SignAC) の支援に感謝する。
また、論文の別刷、コピーあるいはPDFの1部を、ご提出いただければ幸甚です。
御協力の程、よろしくお願い致します。
内規
>内規
