ニュース
2025.2.6
時系列scRNA-seqデータから細胞分化ダイナミクスを推定 – 包括的軌跡推定フレームワークscEGOTの開発
ヒトの発生過程において、細胞がどのように特殊化された細胞型に変化するかを理解することは、生物学における最重要課題の一つです。この複雑なプロセスは「細胞分化」と呼ばれ、発生生物学や再生医療の進歩において重要な鍵を握っています。近年大きく発展した1細胞遺伝子発現解析技術(scRNA-seq)は、1つの細胞が持つすべての遺伝子発現情報を取得する手法で、発生生物学の分野でも広く普及しています。しかしながら、一つの実験から得られる膨大なデータから、目的とする動的現象に関連する情報を正確に取得することは、大きな課題の一つでした。
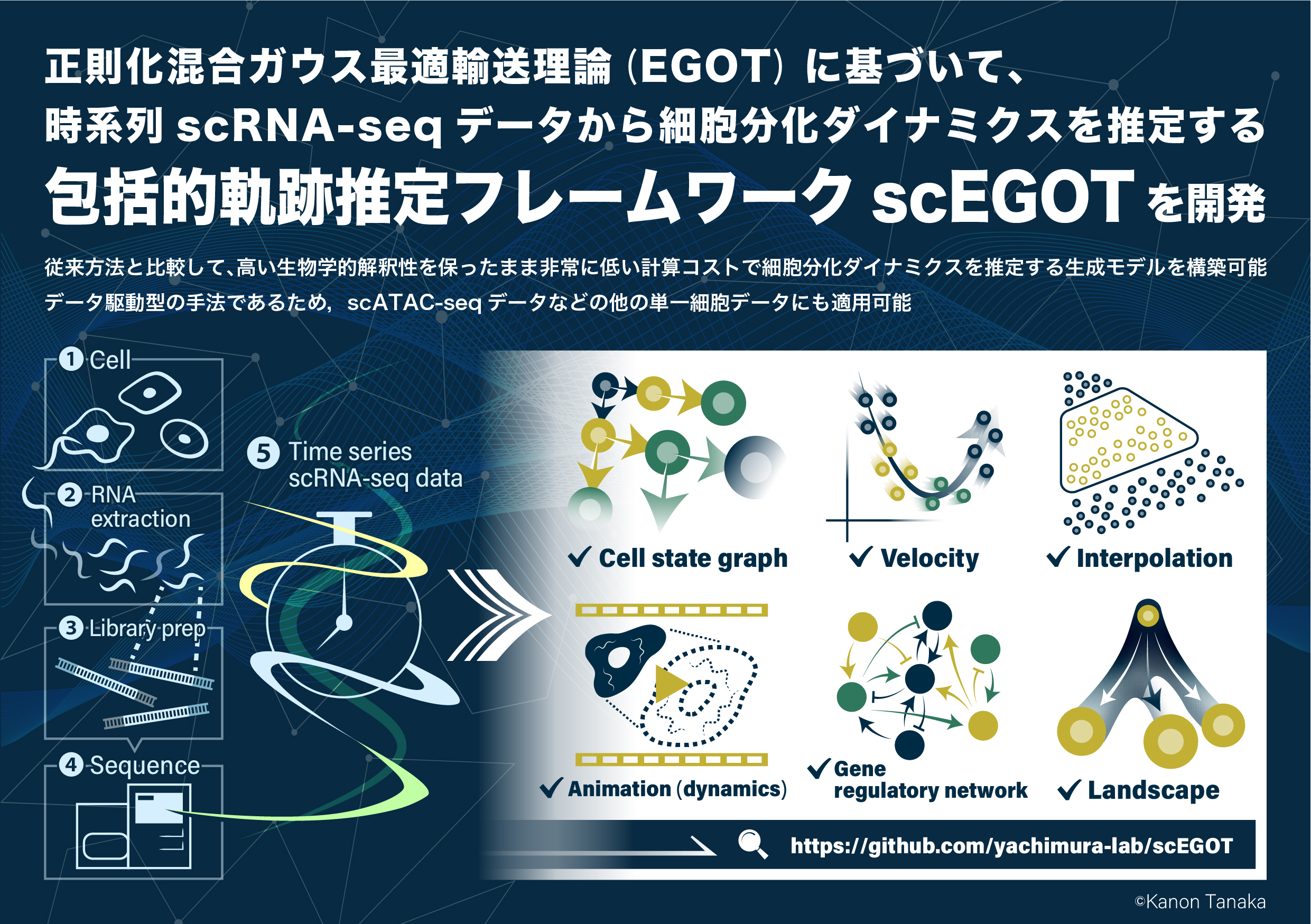
scEGOT概要
京都大学高等研究院ヒト生物学高等研究拠点(WPI-ASHBi)谷地村敏明 特定研究員(研究当時、現:東北大学 数理科学共創社会センター 助教)、平岡裕章 副拠点長/主任研究者らの研究チームは、正則化混合ガウス最適輸送理論に基づく、新しい包括的軌跡推定フレームワーク「single-cell trajectory inference framework based on entropic Gaussian mixture optimal transport(scEGOT)」を開発しました。scEGOTは従来の軌跡予想の手法の限界を克服し、単一細胞が異なる細胞に分化する動的な過程を解析する新たな方法を提供します。
本成果は、2024年12月23日に国際学術誌「BMC Bioinformatics」に掲載されました。
研究者のコメント
scEGOTは多用途なデータ駆動型の手法であり、scRNA-seqに限定されることなく、scATAC-seqなどの他の単一細胞データにも適用可能です。エピジェネティックなメカニズムを解明し、細胞分化や系統決定の理解を深めることで、発生生物学の研究がさらに躍進することを期待しています。(谷地村敏明)
書誌情報
Yachimura, T., Wang, H., Imoto, Y., Yoshida, M., Tasaki, S., Kojima, Y., Yabuta, Y., Saitou, M., & Hiraoka, Y. (2024). scEGOT: single-cell trajectory inference framework based on entropic Gaussian mixture optimal transport. BMC Bioinformatics. https://doi.org/10.1186/s12859-024-05988-z
