ニュース
2022.8.9
1細胞データ解析の精度が飛躍的に向上する前処理法の開発
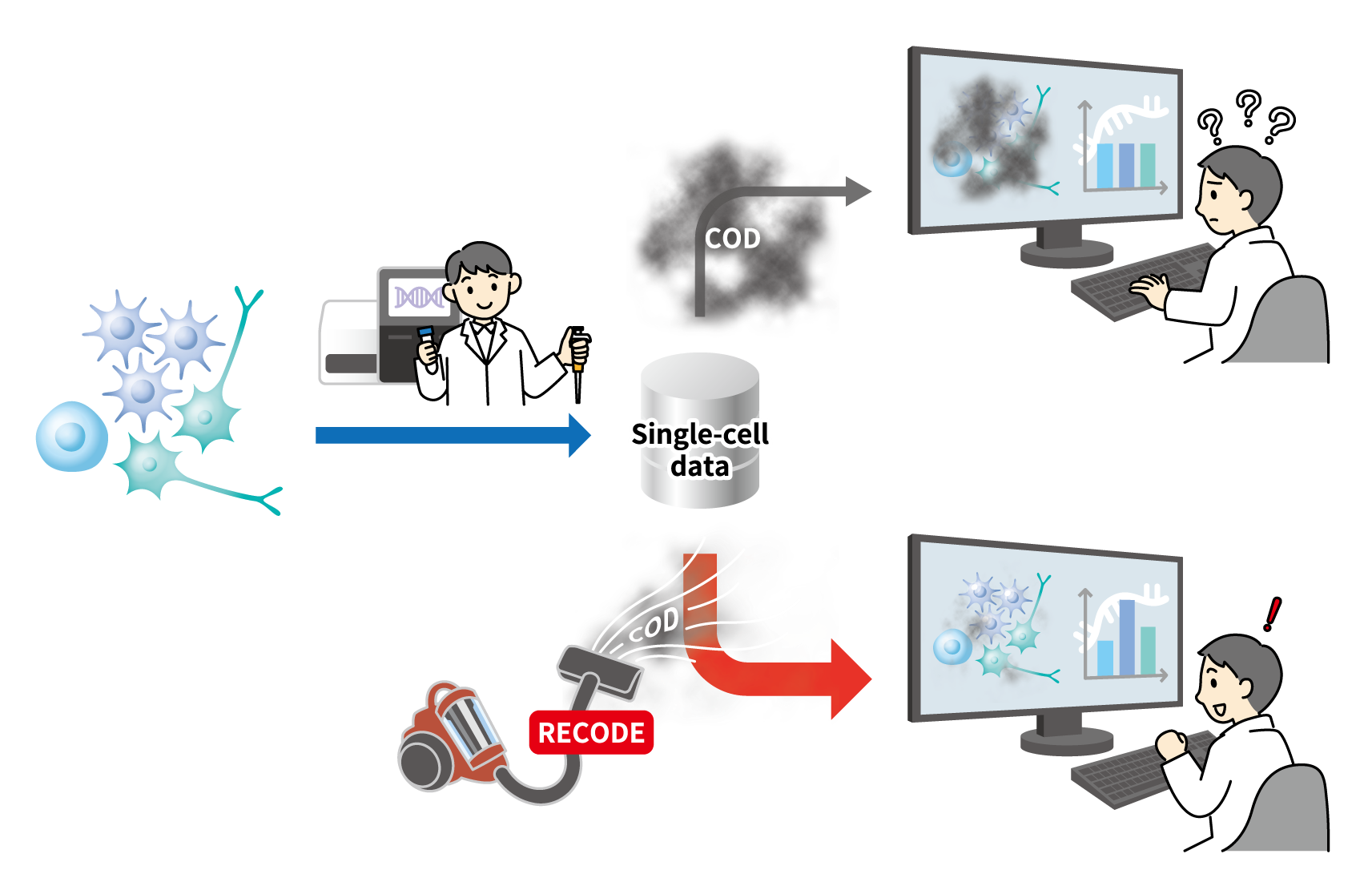
イメージ図 (Credits: TRAIS )
京都大学高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)の井元佑介 特定助教、京都大学 白眉センターおよびASHBiの中村友紀 特定准教授らの研究グループは、高次元統計学という数学理論を用いて、1細胞遺伝子発現データに含まれる非生物学的なノイズを削減する新しい前処理技術「RECODE」を開発しました。これまでの1細胞遺伝子発現データ解析では、非生物学的なノイズにより1細胞レベル解像度の情報を獲得することが困難でしたが、本技術を用いることでデータの質が飛躍的に向上し、高解像度でのデータ解析が可能となりました。RECODEを用いた1細胞遺伝子発現データ解析により、これまで見えなかった生命現象の発見が期待され、ひいては生命科学研究やその臨床応用を発展させると期待されます。本研究成果は、2022年8月9日に『Life Science Alliance』誌で公開されます。
近年、遺伝子解析技術が急速に発達し、短時間で大量の遺伝子情報を取得できるようになりました。なかでも、1細胞遺伝子発現解析は、ひとつひとつの細胞の全遺伝子発現量をデータ化する技術であり、そのデータ解析は、未知の細胞や遺伝子の機能を1細胞レベルで解明する強力な手段として注目を集めています。しかし、実験技術の限界から、実際の数~十数パーセント程度の遺伝子発現量しか検出できず、その検出率の低さが原因で生物学的に正しくないゆらぎ(ノイズ)が観測されてしまいます。さらに、1細胞遺伝子発現データ解析では、数万の遺伝子を同時に扱うため、計算時にノイズが蓄積し、データ解析結果に深刻な影響を及ぼす「次元の呪い」と呼ばれる問題が発生します。
そこで、本研究グループは、高次元統計学という比較的新しい数学理論を用いて、1細胞遺伝子発現データ解析の次元の呪いを解決する前処理技術「RECODE(resolution of the curse of dimensionality)」を開発しました。RECODEを1細胞遺伝子発現データ解析の前処理として利用することで、全てのデータ解析結果が改善され、1~数細胞の特徴づけや低発現遺伝子の機能など、より解像度の高い生物学的情報を獲得できます。さらに、RECODEはどの細胞種にも適用可能で、パラメータが無く、計算が高速で、ノイズ削減の効果を事後評価できるといった実用的な特徴を持っています。
本研究グループは、RECODEを実際の1細胞遺伝子発現データに適用し、次元削減、統計量評価、クラスタリング、遺伝子発現分布、発現変動解析(DEG解析)などのデータ解析結果が改善することを示しました。さらに、RECODEをマウス原腸陥入期の胚発生データに適用し、これまでノイズに埋もれていた希少細胞種やその詳細な分化経路が発見できることを示しました。
本成果により、ノイズに埋もれていた1細胞遺伝子発現データの隠された真実を暴くことができるようになるため、1細胞遺伝子発現データ解析に基づく生命科学研究の可能性を広げることができます。RECODEを用いた1細胞遺伝子発現データ解析は、生命科学のあらゆる分野の新発見に貢献することができ、遺伝子疾患のメカニズムの解明や新薬の開発など、臨床応用の進歩を強力に後押しします。
なお、RECODEの計算プログラム(Python/Rコード、デスクトップアプリケーション)はGitHub(https://github.com/yusuke-imoto-lab/RECODE)で公開されています。
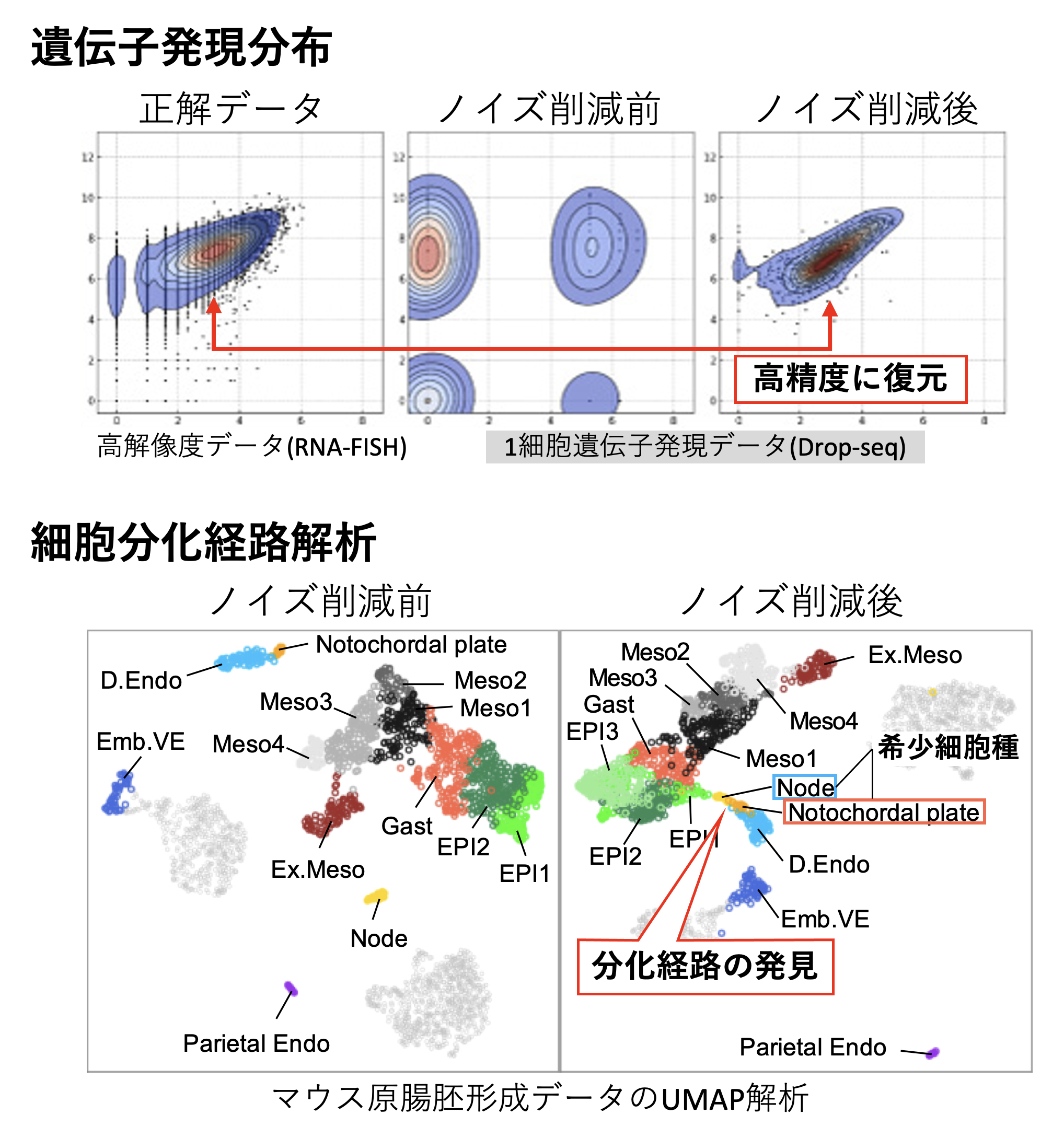
ノイズ削減 の性能比較。(上)遺伝子発現分布の復元。(下)希少細胞種 (Node・Notochordal plate) の同定・細胞分化経路の発見。
論文書誌情報
| タイトル | Resolution of the curse of dimensionality in single-cell RNA sequencing data analysis |
| 著者 | Yusuke Imoto†, Tomonori Nakamura†, Emerson G. Escolar, Michio Yoshiwaki, Yoji Kojima, Yukihiro Yabuta, Yoshitaka Katou, Takuya Yamamoto, Yasuaki Hiraoka, and Mitinori Saitou. †Contributed equally to this work. |
| 掲載誌 | Life Science Alliance |
| DOI | https://doi.org/10.26508/lsa.202201591 |
| 公開日 | 8月9日午後10時(日本時間) |
