ニュース
2022.4.1
世界標準のカニクイザルゲノム配列を確立
創薬に資するゲノミクスデータベースD3Gの更新
D3Gのトップページ
概要
理化学研究所(理研)科技ハブ産連本部予防医療・診断技術開発プログラムの河合純副プログラムディレクターらの共同研究グループ※は、核酸医薬[1]の安全性と有効性の評価において重要なカニクイザル[2]の精度の高い短鎖塩基配列決定データと、CAGE法[3]によりRNA転写開始点に関するデータを新たに取得し、ゲノム塩基配列とRefSeqデータベース[4]の精密化に成功しました。その結果、ゲノム塩基配列は米国国立生物工学情報センター(NCBI)により世界で最も品質の高いデータセットとして認められ、遺伝子研究における標準配列として選定されました。このデータを、ヒト、コモンマーモセット[5]、ラット、マウスのゲノムと遺伝子の最新の情報とともに、創薬に資するゲノミクスデータベース「D3G(Database for Drug Development based on Genome and RNA)[6]」(https://d3g.riken.jp)に追加しました。
本データベースを更新したことで、核酸医薬の開発や薬事における安全性・有効性評価の効率化、創薬における新たなイノベーションにつながると期待できます。また、グローバルに展開される創薬活動において参照する基盤データを、国際標準として発信できたことは非常に有意義です。
D3Gに追加されたデータは、ライフサイエンス統合データベースセンターが運用する塩基配列高速検索サービス「GGGenome」(https://gggenome.dbcls.jp/)や、社内完結型の運用を可能にする高速塩基配列検索ソフトウェア「GGGenomeパッケージ版(創薬パック)」(株式会社レトリバ)からも利用できます。
※共同研究グループ
理化学研究所
科技ハブ産連本部 予防医療・診断技術開発プログラム
副プログラムディレクター 河合 純(かわい じゅん)
生命医科学研究センター
遺伝子制御回路研究チーム
専任研究員 伊藤 昌可(いとう まさよし)
マイクロバイオーム研究チーム
副チームリーダー 須田 亙(すだ わたる)
応用ゲノム解析技術研究チーム
チームリーダー 岡﨑 康司(おかざき やすし)
技師 山本 由美子(やまもと ゆみこ)
予防医療・ゲノミクス応用開発ユニット
特別研究員(研究当時) 廣瀬 直毅(ひろせ なおき)
テクニカルスタッフⅠ(研究当時)佐野 浩美 (さの ひろみ)
(現 理研-IFOMがんゲノミクス連携研究チーム テクニカルスタッフⅠ)
生命機能科学研究センター 分子配列比較解析チーム
チームリーダー 工樂 樹洋(くらく しげひろ)
東京都医学総合研究所 ゲノム医学研究センター
副センター長 川路 英哉(かわじ ひでや)
京都大学 高等研究院ヒト生物学高等研究拠点(WPI-ASHBi)
教授 村川 泰裕(むらかわ やすひろ)
(理化学研究所 生命科学研究センター 理研-IFOMがんゲノミクス連携研究チーム)
国立医薬品食品衛生研究所 遺伝子医薬部
部長 井上 貴雄(いのうえ たかお)
滋賀医科大学 動物生命科学研究センター 幹細胞・疾患モデル研究部門
教授 依馬 正次(えま まさつぐ)
(京都大学 高等研究院ヒト生物学高等研究拠点(WPI-ASHBi)主任研究者)
実験動物中央研究所 マーモセット医学生物学研究部
部長 佐々木 えりか(ささき えりか)
慶應義塾大学 理工学部 生命情報学科
教授 榊原 康文(さかきばら やすぶみ)
研究員 ワサンタン・ジャヤクマル(Vasanthan Jayakumar)
情報・システム研究機構 データサイエンス共同利用基盤施設
ライフサイエンス統合データベースセンター
特任助教内藤 雄樹(ないとう ゆうき)
研究支援
本研究は、日本医療研究開発機構(AMED)ゲノム創薬基盤推進研究事業「RNA標的医薬創出に資する、RNA分子完全長構造に関するデータ基盤の構築」の支援のもと行われました。
1.背景
開発段階の医薬品をヒトへ投与する前に安全性を確認する前臨床試験では、遺伝的にヒトに近い実験動物である非ヒト霊長類を用いた評価が重要です。特にアンチセンス医薬[7]やsiRNA医薬[8]に代表される核酸医薬の開発では、ゲノムや遺伝子の配列情報を考慮した効果・副作用の評価が必要です。共同研究グループはこれまでに、カニクイザルとコモンマーモセットのゲノムアセンブル[9]と遺伝子モデルを構築し、これらを載録したデータベース「D3G(Database for Drug Development based on Genome and RNA)」を公開・運用してきました注1、2)。また、高速塩基配列検索ソフトウェア「GGGenomeパッケージ版」の商品化に協力しました注3)。
今回、創薬におけるデータの信頼性を増すために、カニクイザルのゲノム配列データの精度向上および遺伝子アノテーション[10]の品質向上と標準化に取り組みました。
注1)2020年3月25日理化学研究所お知らせ「創薬に資する霊長類のオミックスデータベースD3Gの公開」
注2)2021年6月30日慶應義塾大学プレスリリース「カニクイザルとコモンマーモセットの全ゲノム配列解読に成功」
注3)2021年4月7日株式会社レトリバプレスリリース「高速塩基配列検索ソフトウェアGGGenomeパッケージ版 創薬基本パック2021年版提供開始」
2.研究手法と成果
共同研究グループは、読み取り精度の高い短鎖塩基配列決定データを用いることで、カニクイザルのゲノムアセンブルの塩基配列の誤り補正を一塩基対単位で実施し、精度の改善に成功しました。その結果は、さまざまな生物種におけるゲノム配列の標準(リファレンス)を選定している米国国立生物工学情報センター(NCBI)のRefSeqデータベースに採用され、世界中の研究者や創薬関係者が標準的に利用するデータとして位置付けられました(図1)。また、共同研究グループが取得したRNA転写開始点[3]に関するCAGEデータ[3]が、高い信頼性を持つ遺伝子モデルの構築に貢献しました。
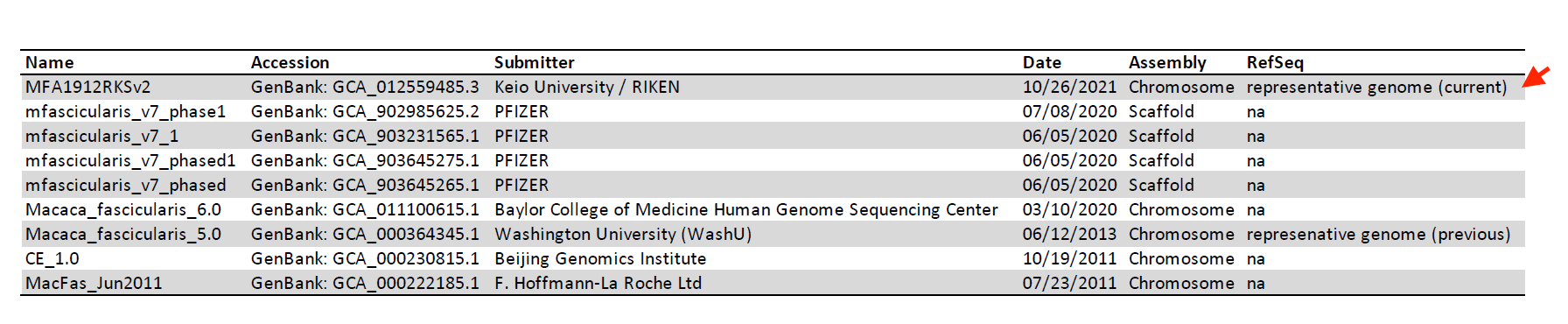
図1 カニクイザルのゲノムアセンブル一覧
NCBI/EMBL-EBI/DDBJに登録されているデータを基に、共同研究グループが作成。共同研究グループが今回構築したMFA1912RKSv2が標準として採用された(赤矢印)。
今回標準として位置付けられた上記のゲノムと、遺伝子モデルの塩基配列をD3Gに加えるとともに、ヒト、コモンマーモセット、ラット、マウスのゲノムと遺伝子モデルの標準データを最新版(Release 22.02)に更新しました(図2)。これらのデータは、ライフサイエンス統合データベースセンターが運用する塩基配列高速検索サービス「GGGenome」や、社内完結型の運用を可能にする高速塩基配列検索ソフトウェア「GGGenomeパッケージ版(創薬パック)」(株式会社レトリバ)にも収録されました。

図2 D3Gのトップページ
3.今後の期待
本成果は医薬品の安全性評価、特に核酸医薬の安全性評価に大きく貢献するとともに、医科学や生物学などの基礎研究の基盤データを提供します。特に創薬における安全性と有効性の効果的かつ公正な評価には、最新の科学技術で整備された標準データが欠かせません。グローバルに展開される創薬活動において参照することできるカニクイザルの基盤データを、国際標準として発信できたことは非常に有意義です。今後もアカデミア、製薬企業、規制当局らが協力を継続し、さらにデータを充実させるとともに、疾患や遺伝子機能に関する情報を連携させることが期待されます。
4.補足説明
- 1.核酸医薬
一般に、核酸あるいは修飾核酸が十数~数十塩基連結したオリゴ核酸で構成され、タンパク質に翻訳されることなく直接生体に作用するもので、化学合成により製造される医薬品を指す。タンパク質を標的とする低分子医薬や抗体医薬による創薬シーズの枯渇が危惧されるなか、核酸医薬はRNAを標的とした創薬が可能であることなどから、次世代のモダリティとして注目を集めている。 - 2.カニクイザル
旧世界ザルに属し、主に東南アジアに生息する。ヒトに最も近縁な実験動物として、ワクチンや医薬品の安全性試験に長らく用いられてきた。近年は遺伝子改変技術が進展してヒト病態モデルの作製が簡便になっており、薬効試験への適用が期待されている。 - 3.CAGE法、転写開始点、CAGEデータ
CAGE法はRNA分子の5´端の数十塩基を次世代シーケンサーで大規模に解読する手法。CAGE法によって、RNAの転写が開始される位置(転写開始点)を特定できる。また、CAGE法によって解読された5´端の数十塩基をCAGEデータと呼び、その数を計測することで、遺伝子の転写量を取得できる。CAGE法は国際プロジェクトFANTOMで利用されている。CAGEはCap-analysis of gene expressionの略。 - 4.RefSeqデータベース
Reference Sequence Databaseの略。遺伝子解析の基準として参照するための遺伝子配列データベースとして米国国立生物工学情報センター(NCBI)が構築している。世の中に発表される多くの遺伝子情報の中から、代表的な配列としてふさわしい品質の高いものが選ばれ、注釈を付されている。 - 5.コモンマーモセット
ヒトと同じ霊長類に属する新世界ザルの一種で、南アメリカ・ブラジル大西洋岸の熱帯・亜熱帯林に生息する。高い繁殖能力を有し、人獣共通感染症を持たないなどの多数の利点から、実験動物としてパーキンソン病、アルツハイマー病などのヒト病態モデルが開発されている。 - 6.D3G(Database for Drug Development based on Genome and RNA)
創薬や医科学研究に有用なヒトや霊長類、モデル動物の全ゲノム配列や遺伝子モデル、毒性評価試験の対象とされる組織の遺伝子発現情報を載録した統合データベース。RNAを標的とする核酸医薬の創薬や、モデル動物のゲノム編集などにおいて広く利用されている。 - 7.アンチセンス医薬
標的RNAと相補的に結合することで機能する核酸医薬で、修飾核酸を含む1本鎖DNAで構成される。RNA分解やスプライシング制御などの作用機序で有効性を発揮する。 - 8.siRNA医薬
標的mRNAと相補的に結合し、RNA干渉(RNAi)の原理でmRNAを切断する核酸医薬。20塩基長程度の2本鎖RNAで構成される。 - 9.ゲノムアセンブル
ゲノムDNAを塩基配列解析装置で解析し得られる大量のDNA断片の塩基配列から再構築されたゲノムの長い塩基配列。塩基配列の質や量、再構築に用いられるバイオインフォマティクス技術の性能により、再構築されたゲノム配列の質は変化する。 - 10.遺伝子アノテーション
ゲノムなどの特定の塩基配列や領域に付与される、その特徴や機能の注釈。実験の実データだけでなくコンピュータによる類推結果も加味される。関連するライフサイエンス研究や医療、創薬などにおける利用のために、高い品質の遺伝子アノテーションが求められる。
5.謝辞
本研究を進める上で多くの方からご協力をいただきました。特に以下の方々にあつく感謝します(敬称略)。
日本製薬工業協会医薬品評価委員会 基礎研究部会 渡部一人、木下潔、太田哲也
日本核酸医薬学会レギュラトリーサイエンス部会
理化学研究所 ピエロ・カルニンチ、粕川雄也
独立行政法人 医薬品医療機器総合機構 毒性領域
株式会社レトリバ 河原一哉、億田正貴
6.発表者
(2022年3月31日まで)
理化学研究所 科技ハブ産連本部 予防医療・診断技術開発プログラム
副プログラムディレクター 河合 純(かわい じゅん)
(2022年4月1日から)
地方独立行政法人神奈川県立産業技術総合研究所 研究開発部
常勤研究員 河合 純(かわい じゅん)
