
薮田 幸宏
研究者 (斎藤G)
- Position
- 助教
- Research Field
- バイオインフォマティクス、発生生物学、分子生物学
研究概要
ヒト、カニクイザル、マウスの生殖細胞の発生におけるエピゲノムの解析
哺乳類の生殖細胞は、胎児期の発生過程において次世代への全能性の再獲得のためにエピゲノムのリプログラミングを行ってゲノムを初期化しながら精子や卵に分化する。この過程のエピゲノムの変化を調査することは、ゲノムおよびエピゲノム情報がどの様に次世代に引き継がれるのか、また、この過程における異常がどの様な疾患を引き起こす可能性があるのかを調査するのに重要である。また、この過程が生物種間でどの程度保存されているのかを調べることによって、どの程度モデル生物における研究が役に立つのかを予見できる可能性がある。
近年、当ASHBi斎藤グループにおいてマウスES細胞、iPSC細胞から、正常な精子や卵に分化できる始原生殖細胞様細胞(PGCLC)を試験管内で誘導することに成功し、続いてヒトiPS細胞から、さらにカニクイザルES細胞およびiPS細胞からPGCLCを誘導することに成功した。これらPGCLCによって、細胞数が少ないために不可能だった生殖細胞に対するChIP-seqやBisulfite-seqといったエピゲノム解析が可能になった。
マウスおよびヒトのPGCLCのBisulfite-seq解析から、マウスではPGCLC誘導によって、ヒトではその後の卵原細胞誘導によってゲノム全体のDNAの脱メチル化が誘導され、生体内の生殖細胞と同様の現象を確認した。しかし、その脱メチル化にかかる期間は、マウスとヒトでは大きく異なり、マウスでは十日以内にほぼゲム全体が脱メチル化されるが、ヒトでは120日以上を必要とした。どちらの生物種でも特定のレトロトランスポゾンの領域では脱メチル化に抵抗性がみられ、レトロトランスポゾンによるDNAへのダメージを回避するメカニズムが考えられる。この様にヒトとマウスの脱メチル化パターンには共通な部分と異なる部分が存在し、そのメカニズムはまだ解明されていない。本研究では、さらにカニクイザルのゲノムの脱メチル化も比較しながら、そのメカニズムを解明する。
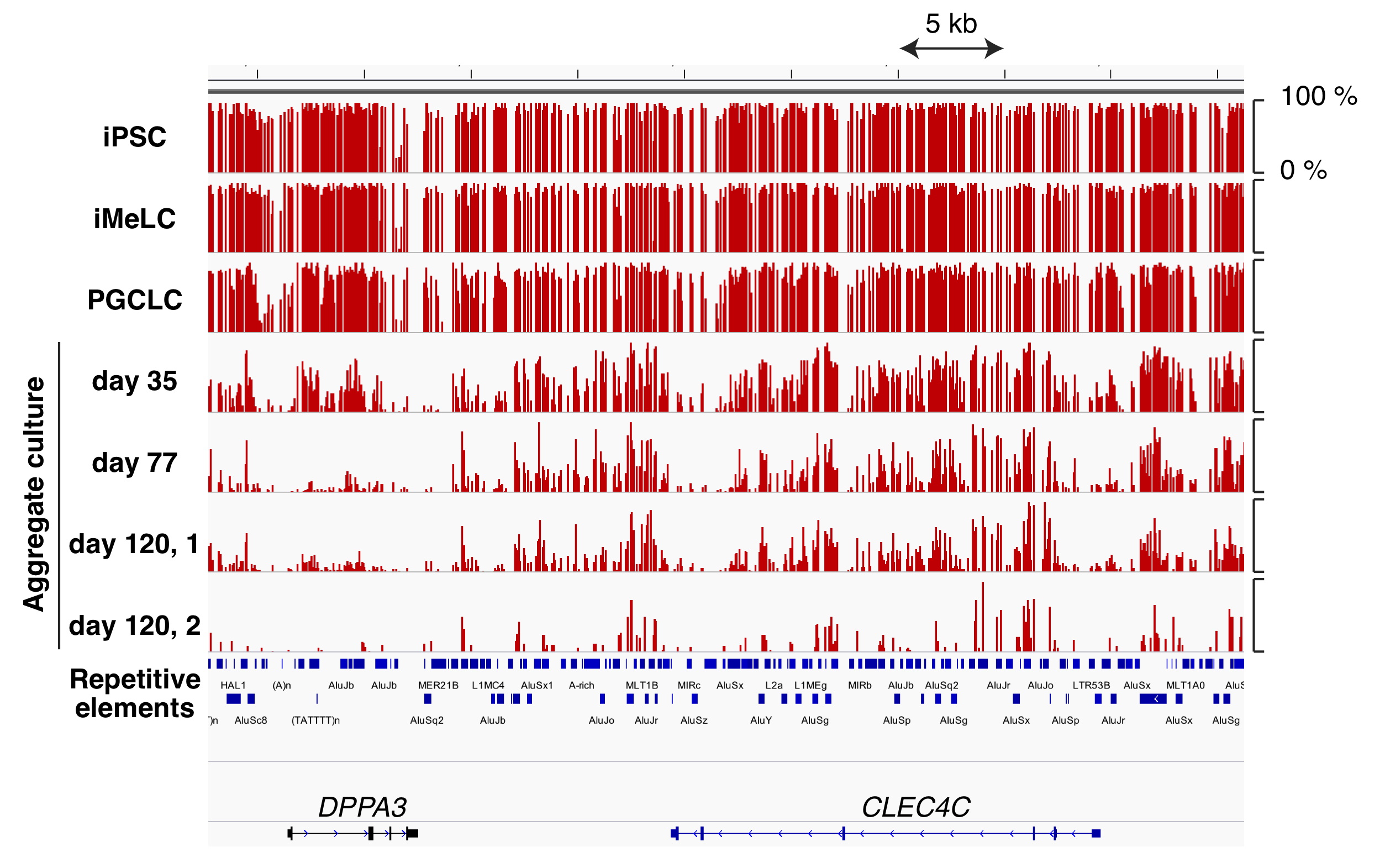
図1. ヒトiPSC細胞から卵原細胞を誘導する過程において、ゲノム全体でDNAの脱メチル化が起こる。
略歴
2002年 福井県立大学大学院生物資源学研究科 博士後期課程終了。2002年―2003年 米国オレゴンヘルス&サイエンス大学生化学部にてポスドク研究員。2003年―2010年 理化学研究所神戸研究所発生・再生科学総合研究センター(CDB)にてポスドク研究員。2010年―2019年 京都大学医学研究科にてポスドク研究員。2019年より現職。
論文
Nagaoka SI, Nakaki F, Miyauchi H, Nosaka Y, Ohta H, Yabuta Y, Kurimoto K, Hayashi K, Nakamura T, Yamamoto T, Saitou M. ZGLP1 is a determinant for the oogenic fate in mice. Science. 2020 Mar 6;367(6482).
Sakai Y, Nakamura T, Okamoto I, Gyobu-Motani S, Ohta H, Yabuta Y, Tsukiyama T, Iwatani C, Tsuchiya H, Ema M, Morizane A, Takahashi J, Yamamoto T, Saitou M. Induction of the germ cell fate from pluripotent stem cells in cynomolgus monkeys. Biol Reprod. 2020 Mar 13;102(3):620-638.
Yamashiro C, Sasaki K, Yabuta Y, Kojima Y, Nakamura T, Okamoto I, Yokobayashi S, Murase Y, Ishikura Y, Shirane K, Sasaki H, Yamamoto T, Saitou M. Generation of human oogonia from induced pluripotent stem cells in vitro. Science. American Association for the Advancement of Science; 2018 Oct 19;362(6412):356–60.
Mitani T, Yabuta Y, Ohta H, Nakamura T, Yamashiro C, Yamamoto T, Saitou M, Kurimoto K. Principles for the regulation of multiple developmental pathways by a versatile transcriptional factor, BLIMP1. Nucleic Acids Res. 2017 Dec 1;45(21):12152–69.
Miyauchi H, Ohta H, Nagaoka S, Nakaki F, Sasaki K, Hayashi K, Yabuta Y, Nakamura T, Yamamoto T, Saitou M. Bone morphogenetic protein and retinoic acid synergistically specify female germ-cell fate in mice. EMBO J. EMBO Press; 2017 Nov 2;36(21):3100–19.
Kojima Y, Sasaki K, Yokobayashi S, Sakai Y, Nakamura T, Yabuta Y, Nakaki F, Nagaoka S, Woltjen K, Hotta A, Yamamoto T, Saitou M. Evolutionarily Distinctive Transcriptional and Signaling Programs Drive Human Germ Cell Lineage Specification from Pluripotent Stem Cells. Cell Stem Cell. 2017 Oct 5;21(4):517–532.e5.
Ohta H, Kurimoto K, Okamoto I, Nakamura T, Yabuta Y, Miyauchi H, Yamamoto T, Okuno Y, Hagiwara M, Shirane K, Sasaki H, Saitou M. In vitro expansion of mouse primordial germ cell-like cells recapitulates an epigenetic blank slate. EMBO J. EMBO Press; 2017 Jul 3;36(13):1888–907.
Nakamura T, Yabuta Y, Okamoto I, Sasaki K, Iwatani C, Tsuchiya H, Saitou M. Single-cell transcriptome of early embryos and cultured embryonic stem cells of cynomolgus monkeys. Sci Data. 2017 Jun 20;4:170067.
Yokobayashi S, Okita K, Nakagawa M, Nakamura T, Yabuta Y, Yamamoto T, Saitou M. Clonal variation of human induced pluripotent stem cells for induction into the germ cell fate. Biol Reprod. 2017 Jun 1;96(6):1154–66.
Honda A, Kawano Y, Izu H, Choijookhuu N, Honsho K, Nakamura T, Yabuta Y, Yamamoto T, Takashima Y, Hirose M, Sankai T, Hishikawa Y, Ogura A, Saitou M. Discrimination of Stem Cell Status after Subjecting Cynomolgus Monkey Pluripotent Stem Cells to Naïve Conversion. Sci Rep. 2017 Mar 28;7(1):487.
研究グループ
斎藤グループ着任日
2019年2月1日
