ニュース
2020.10.26
エンハンサーの機能を効率的に大規模解析する技術を確立
ゲノムの機能、病気、進化などの高次生命現象の理解への貢献に期待
研究の概要図
概要
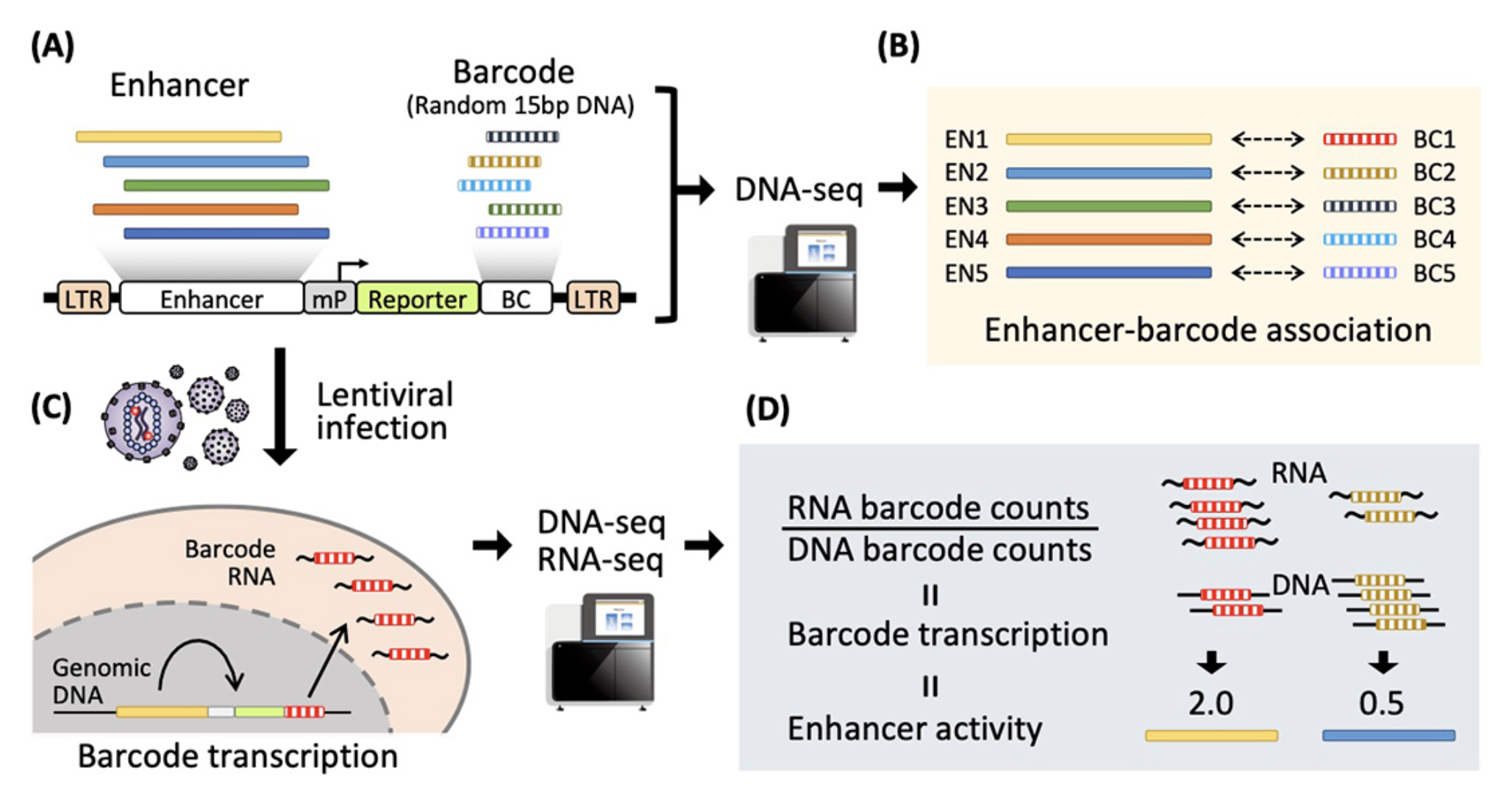
私たちのゲノム(DNAに含まれる全遺伝情報)のうちタンパク質をコードしている遺伝子DNAはわずか1.5%であり、98%以上はノンコーディングDNA(注1)が占めています。ノンコーディングDNA領域にはエンハンサー(注2)と呼ばれる遺伝子発現制御エレメントが点在しており、その変異は病気や発生異常の原因となります。しかしながら、広大なゲノムDNAの中からエンハンサーを同定し、その機能を解析する事は容易ではありません。近年、エンハンサーの機能を大規模並列的に定量解析できる新規技術、大規模並列レポーターアッセイ法 (MPRA)(注3)が開発され、ゲノム中のエンハンサー機能が急速に解ってきています。ところが、MPRAは本研究グループをはじめ複数の研究室で独立に開発されており、異なる実験デザインが採用されていました。本研究において同グループは、ベクター(注4)の構造、エンハンサーの位置、向き、長さなどの要素について様々な比較検討を行うことで、MPRAの洗練と確立を試みました。これにより、エンハンサーの効率的な大規模機能解析が可能となり、ゲノムの機能や、病気、進化などの高次生命現象の理解に繋がると期待されます。
本研究成果は、2020年10月12日に英国の国際学術誌「Nature Methods」に掲載されました。
研究プロジェクトについて
本研究は、カリフォルニア大学サンフランシスコ校(UCSF)の井上詞貴 博士研究員(研究当時、現:京都大学高等研究院 ヒト生物学高等研究拠点(WPI-ASHBi)特定准教授)、Nadav Ahituv同教授およびワシントン大学のJay Shendure教授らの共同研究グループによる成果であり、NIH(米国立衛生研究所)およびNHGRI(米国立ヒトゲノム研究所)等の支援を受けて行われました。用語解説
注1) ノンコーディングDNA:真核生物ゲノムの大部分を占める、タンパク質をコードしないDNA領域。マイクロRNA、非翻訳領域や、プロモーター・エンハンサーといった転写制御エレメントなどが含まれる。
注2) エンハンサー:細胞内外のシグナルやストレスに応答し、転写因子の結合やヒストンの修飾を介して、遺伝子が「いつ」「どこで」「どのくらい」発現するかを厳密に調節するDNA配列。
注3) 大規模並列レポーターアッセイ法:Massively Parallel Reporter Assay (MPRA)。数千から数万のエンハンサーの機能を、1度の実験で大規模並列的に定量解析することが出来る新技術。15塩基対のランダム配列(バーコードと呼ぶ)をエンハンサーに連鎖させ、次世代シーケンサーを用いてバーコード転写量を定量することにより、これを実現する。
注4) ベクター:人工DNAを培養細胞などに導入するための分子生物学ツール。プラスミドやレンチウィルスなど。
研究者のコメント
近年ゲノム科学が急速に発展し、医療や基礎生物学に関する様々な知見が得られていますが、新発見には新しい実験手法・技術の開発が不可欠です。本研究は、実験技術の丁寧な比較検討を報告することで後の新発見を縁の下から支えるものであり、実際にこの技術を駆使して得た進化や疾患に関連する様々な研究結果を報告する予定です。今後の研究発展にご期待下さい。論文書誌情報
- タイトル
A systematic evaluation of the design and context dependencies of massively parallel reporter assays(大規模並列レポーターアッセイ法実験条件の体系的検証) - 著 者
Jason C. Klein, Vikram Agarwal, Fumitaka Inoue, Aidan Keith, Beth Martin, Martin Kircher, Nadav Ahituv & Jay Shendure - 掲 載 誌
Nature Methods - DOI
https://doi.org/10.1038/s41592-020-0965-y
