新技術e2MPRA: 遺伝子発現制御原理の解明へ前進
遺伝子スイッチのエピジェネティック状態を大規模並列的に理解する
京都⼤学⾼等研究院ヒト⽣物学⾼等研究拠点 (WPI-ASHBi) 張 子聡研究員、井上 詞貴特定准教授らの研究グループは、シス制御配列における変異が転写活性およびエピジェネティック状態1に与える影響を同一サンプルで大規模並列に定量する新技術を開発しました。
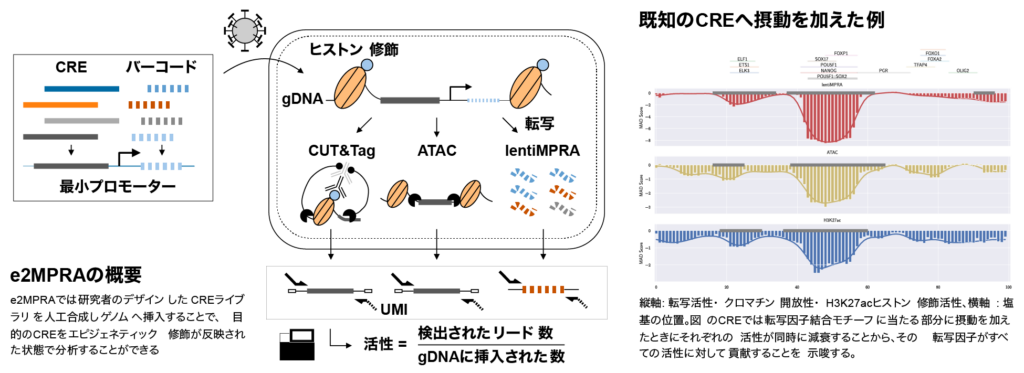
DNAには私たちの「部品」である遺伝子の他に、その遺伝子を「いつ・どこで・どれだけ」使うのかを指示するシス制御配列(cis-regulatory element; 以下CRE)が記されています。遺伝子の配列は個人間でほとんど変わりませんが、CREはより多くの変異を持ち、その違いは個人間の姿形の違いや疾患リスクの主要因の一つになります。CREは転写因子2の足場となり、DNAのエピジェネティック状態を変えることで遺伝子の発現量を調節します。そのためCREのエピジェネティック状態を計測することでCREがどのようなメカニズムで遺伝子発現を調節し、変異によってその機能がどのように変化するのかを検出することができます。しかし、既存のエピジェネティック状態の計測手法はゲノム全体のデータを取得するのを得意とする一方、変異の影響を調べるには多くの労力が必要でした。そこで本研究では、CREの変異の影響を大規模に計測できる新手法enrichment followed by epigenomic profiling MPRA (e2MPRA)を開発しました。提案手法の実証試験において設計した約1万個のCREの転写活性・クロマチン開放性・H3K27acヒストン修飾活性を計測した結果、それぞれのエピジェネティック状態における変異の影響に加え転写因子同士の役割分担や協同効果など、転写制御における様々な法則性を明らかにしました。
本成果は、2026年1⽉14⽇(英国時間)にAccelerated Article Preview版が、2⽉17⽇(⽇本時間2⽉18⽇)に校正版が国際学術誌「Nature Communications」に掲載されました。
用語解説
- エピジェネティック状態: DNAを読むための目印。DNAは普段ヒストンというタンパク質に巻きつきクロマチンと呼ばれる構造をとっている。この目印がついている部分のDNAは解かれたり(クロマチン開放)凝縮したり(クロマチン凝集)して高次の構造を取る。DNAが解かれた部分には転写因子などのタンパク質などが結合でき、機能部位として使用される。対して、DNAが凝縮した部分は機能しなくなる。さらにDNAはアセチル化(H3K27ac)やメチル化などの化学修飾によってもその機能が調整されている。体の細胞はほぼすべての同じDNAを持つが、エピジェネティック修飾が異なることで皮膚や神経、筋肉などの様々な細胞となる。 ↩︎
- 転写因子: DNAの特定の配列(結合モチーフ)に結合して遺伝子の転写がどれだけ起きるのかを調節するタンパク質。多くの場合シス制御配列中に存在し、RNA転写酵素や他の因子が転写を開始しやすくしたり逆にそれらを阻害する役割を持つ。シス制御配列内における転写因子同士の力関係でそのシス制御配列がどれくらい強く遺伝子を駆動するのかが決まる。 ↩︎
研究者のコメント
2022年にようやくヒトゲノムは全配列が解読されました。ヒトゲノム解読後の課題は、個体間の多様な配列差が遺伝子発現や表現型の違いへ至る分子機構を理解することです。本研究で開発したe2MPRAは、シス制御配列の変異が転写活性とエピジェネティック状態に及ぼす影響を同一条件下で並列に定量できるので、個人差や疾患リスクの分子機構を解明するための基盤技術となることを期待します。(張子聡)
書誌情報
Zhang, Z., Georgakopoulos-Soares, I., Bourque, G., Ahituv, N., & Inoue, F. (2026). Simultaneous epigenomic profiling and regulatory activity measurement using e2MPRA. Nature Communications. https://doi.org/10.1038/s41467-026-68422-3