
井上 詞貴
| Position | 准教授 |
|---|---|
| 研究グループ | Bourqueグループ |
| Research Field | ゲノミクス、分子発生学 |
| 受賞 | ENCODEコンソーシアムミーティング ポスター賞(2018) |
| ORCID | https://orcid.org/0000-0003-0657-434X |
| Personal Website | https://sites.google.com/view/inouelabwebsite/home |
| 着任日 | 2020年7月1日 |
研究概要
細胞分化・個体発生・進化に関わるレギュロームの理解
私たちのゲノムのうちタンパク質をコードしている遺伝子DNAはわずか1.5%であり、98%以上はノンコーディングDNAが占めています。ノンコーディングDNA領域には、「エンハンサー」と呼ばれる転写制御エレメントが点在しています。エンハンサーは、細胞内外のシグナルやストレスに応答し、転写因子の結合やヒストンの修飾を介して、遺伝子が「いつ」「どこで」「どのくらい」発現するかを厳密に調節しています。しかしながら、広大なゲノムDNAの中からエンハンサーを同定し、機能解析する事は容易ではなく、ゲノム中の制御エレメントの全体像(レギュローム)は不明のままです。
私たちはこれまで、RNA-seq、ChIP-seq、Cut&Tag、ATAC-seqといったゲノム・エピゲノム解析技術を用いて、細胞分化・個体発生・進化に関わるエンハンサーの同定を試みてきました。さらに、Lentivirus-based massively parallel reporter assay (lentiMPRA)を開発し、エンハンサーの大規模並列的かつ定量的な機能解析を実現しました。lentiMPRAは、転写バーコードを用いることで、数千から数万のエンハンサーのクロマチン環境での活性をハイスループットに解析できる新技術です(図1)。
私たちは、ヒトやチンパンジーの幹細胞から誘導した神経前駆細胞をモデルとし、lentiMPRAにより多数の神経関連エンハンサーを解析しています。そして上述のような最先端ゲノム・エピゲノム解析技術を駆使し、またiPS由来分化細胞やシングルセルテクノロジーと組み合わせながら、細胞分化・個体発生・進化など高次生命現象に関わるレギュロームの理解を目指します。
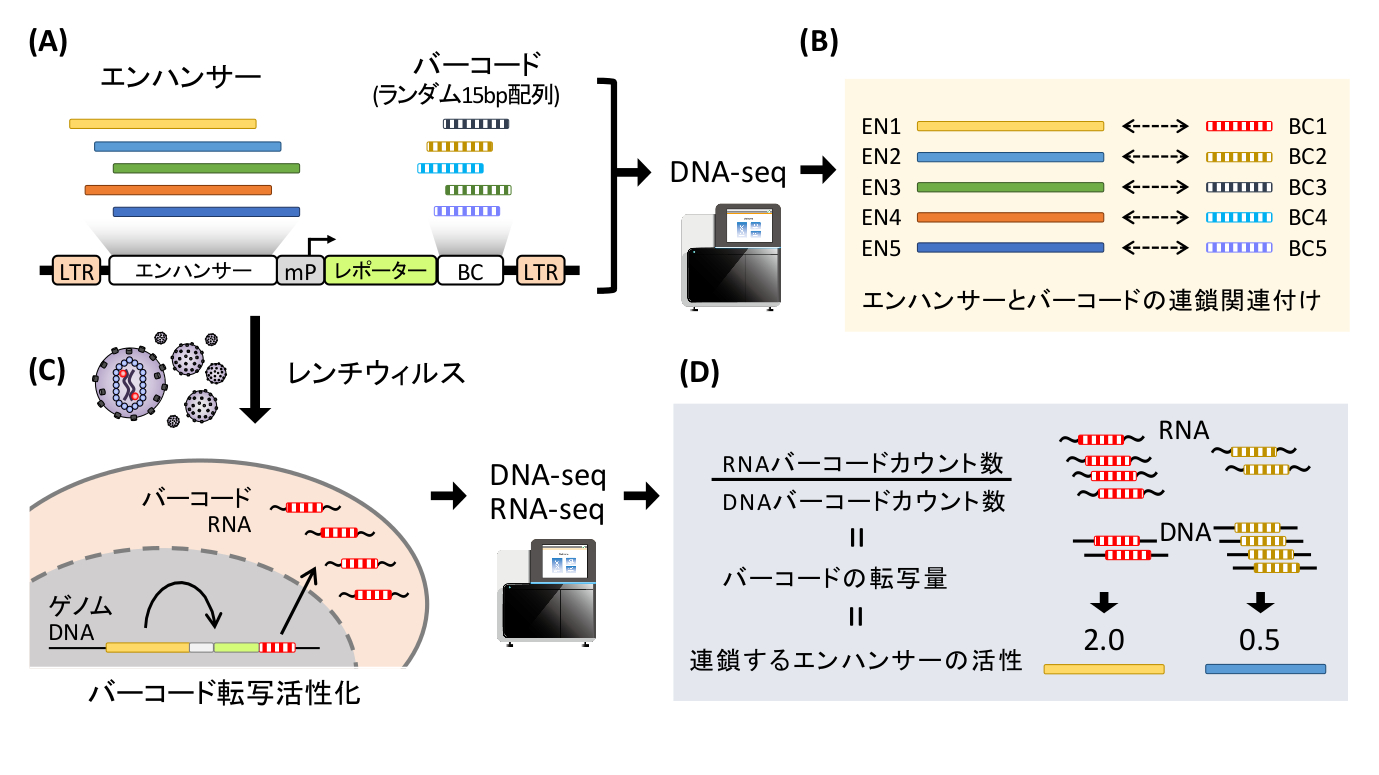
(A) 数千のエンハンサー候補配列を最小プロモーター (mP)の上流へ、バーコード(BC, 15bpランダム配列)をレポーター遺伝子3’UTRへ挿入したライブラリを作成する。(B)ライブラリ内のエンハンサーとバーコードの連鎖をあらかじめDNA-seqにより決定する。(C)レンチウィルスによりライブラリを細胞へ導入する。細胞内ではベクターがゲノムDNAへ挿入され、エンハンサー活性に従ってバーコードが転写される。(D)RNA-seqにより転写バーコードを定量(導入DNAバーコード量で標準化)することで、連鎖するエンハンサーの活性を大規模並列的に解析することができる。
略歴
2008年 埼玉大学大学院理工学研究科 博士後期課程修了。2008年-2012年 理化学研究所 発生・再生科学総合研究センター 研究員。2012年-2020年 カリフォルニア大学サンフランシスコ校 (UCSF) ポストドクトラルフェロー/研究員。2020年より京都大学ヒト生物学高等研究拠点 (ASHBi) 特定拠点准教授。
論文
Kathleen C. Keough, Sean Whalen, Fumitaka Inoue, Pawel F. Przytycki, Tyler Fair, Chengyu Deng, Marilyn Steyer, Hane Ryu, Kerstin Lindblad-Toh, Elinor Karlsson, Zoonomia Consortium, Tomasz Nowakowski, Nadav Ahituv, Alex Pollen, Katherine S. Pollard
Three-dimensional genome rewiring in loci with human accelerated regions
Science 380(6643). (2023)
Sean Whalen*, Fumitaka Inoue*, Hane Ryu*, Tyler Fair, Eirene Markenscoff-Papadimitriou, Kathleen Keough, Martin Kircher, Beth Martin, Beatriz Alvarado, Orry Elor, Dianne Laboy Cintron, Alex Williams, Md. Abul Hassan Samee, Sean Thomas, Robert Krencik, Erik M. Ullian, Arnold Kriegstein, John L. Rubenstein, Jay Shendure, Alex A. Pollen, Nadav Ahituv^, Katherine S. Pollard^
Machine learning dissection of human accelerated regions in primate neurodevelopment
Neuron 111(6), 857-873. (2023)
Anat Kreimer*^, Tal Ashuach*, Fumitaka Inoue*, Alex Khodaverdian, Chengyu Deng, Nir Yosef^, Nadav Ahituv^
Massively parallel reporter perturbation assays uncover temporal regulatory architecture during neural differentiation
Nature communications 13(1), 1504. (2022)
Carly V Weiss*, Lana Harshman*, Fumitaka Inoue, Hunter B Fraser, Dmitri A Petrov^, Nadav Ahituv^, David Gokhman^
The cis-regulatory effects of modern human-specific variants
eLife 10. (2021)
Jason C. Klein*, Vikram Agarwal*, Fumitaka Inoue*, Aidan Keith*, Beth Martin, Martin Kircher, Nadav Ahituv^, Jay Shendure^
A systematic evaluation of the design and context dependencies of massively parallel reporter assays
Nature Methods 17, 1083-1091.(2020)
*co-first authors
^co-corresponding authors


