
Sungrim Seirin-Lee(李 聖林)
| Position | 教授 |
|---|---|
| 研究グループ | Seirinグループ |
| Research Field | 数理生命医学・数理モデル学・応用数理 |
| 受賞 | 現象数理学三村賞(2020) |
| ORCID | https://orcid.org/0000-0002-8466-8037 |
| Personal Website | https://ashbi.kyoto-u.ac.jp/bimed-math/ |
| 着任日 | 2021年10月1日 |

|
研究概要
ヒト発生仕組みにおけるパターン形成の数理的解明及び形と自己免疫性疾患を繋ぐ新しいフュージョンアプローチの構築
近年、数学の力を応用し、様々な現象や問題を解決していく異分野融合研究が大きな成長を成し遂げ、時代を変えようとしています。我々のグループではまさにこのような最先端の応用数理研究の中でもパターン形成に関わる生命科学の発生プロセスの解明と難治性自己免疫疾患における難題の解決に取り組んでいます。
特に、我々人間はどう生まれるのか?という本質に迫る生命科学の謎を数理モデル駆動手法(Model driven approach)とデータ駆動手法(Data driven approach)の融合手法を用いて解明しています。また、生物実験自体が難しく、その解決の手法が極めて限られている臨床医学の課題においては、奇抜な発想転換を用いた数理モデリングを屈指し、「かたち」という概念から臨床医学と細胞生物実験を繋ぐ新しい融合研究手法を構築しています。
我々のグループでは、「パターンと形(かたち)」をキーワードに様々な生命現象の謎を解き明かすことで、細胞の機能制御における新しい概念を生み出し、再生医療や疾患治療に応用することを目指しています。さらに、数理モデルから導かれる生命の普遍性(真理)を発見することで、ヒト生物学の根幹にある原理を解明し、数理ヒト生物学を開拓することを目指しています。
略歴
2010年岡山大学大学院環境学研究科にて博士後期課程早期修了。博士課程在学中にJSPS DC1に採用され、Oxford大学Mathematical Instituteに留学し、パターン形成の数理モデリングを本格的に学ぶ。JSPS PDで東京大学数理科学研究科、理化学研究所を経て、2014年から広島大学理学研究科にて、助教、准教授、2016年―2019年 JSTさきがけの兼任を経て、2020年同大学統合生命科学研究科の教授。2021年10月より京都大学高等研究院・教授、2023年1月より同大学医学研究科・教授(兼任)となり現在に至る。
論文
Seirin-Lee, S^., & Kimura, A^. (2025). Geometric factors for cell arrangement: How do cells determine their position in vivo? Seminars in Cell & Developmental Biology, 169, 103604. https://doi.org/10.1016/j.semcdb.2025.103604
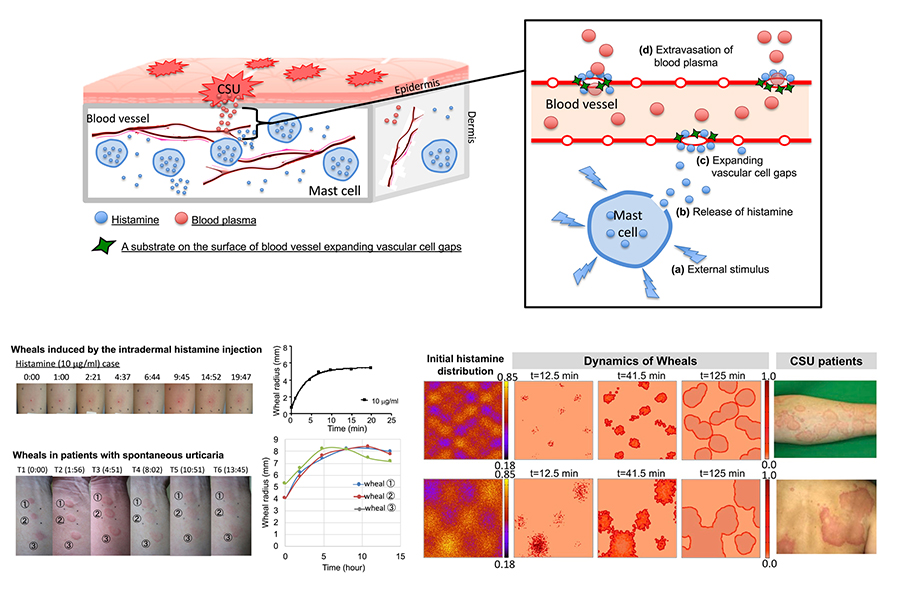
Seirin-Lee, S^., Takahagi, S., & Hide, M^. (2025). Pathophysiological mechanisms of the onset, development, and disappearance phases of skin eruptions in chronic spontaneous urticaria. Bulletin of Mathematical Biology, 87(1). https://link.springer.com/article/10.1007/s11538-024-01380-3
Seirin-Lee, S^., Matsubara, D., Yanase, Y., Kunieda, T., Takahagi, S^., & Hide, M. (2023). Mathematical-based morphological classification of skin eruptions corresponding to the pathophysiological state of chronic spontaneous urticaria. Communications Medicine, 3, 171. https://doi.org/10.1038/s43856-023-00404-8
Seirin-Lee, S^., Yamamoto, K., & Kimura, A^. (2022). The extra-embryonic space and the local contour are critical geometric constraints regulating cell arrangement. Development, 149, dev200401. https://doi.org/10.1242/dev.200401
Seirin-Lee, S. (2021). The role of cytoplasmic MEX-5/6 polarity in asymmetric cell division. Bulletin of Mathematical Biology, 83, 29. https://doi.org/10.1007/s11538-021-00860-0
Seirin-Lee, S., Yanase, Y., Takahagi, S., & Hide, M. (2020). Multifarious eruptions of urticaria solved by a simple mathematical equation. PLOS Computational Biology, 16(1), e1007590. https://doi.org/10.1371/journal.pcbi.1007590
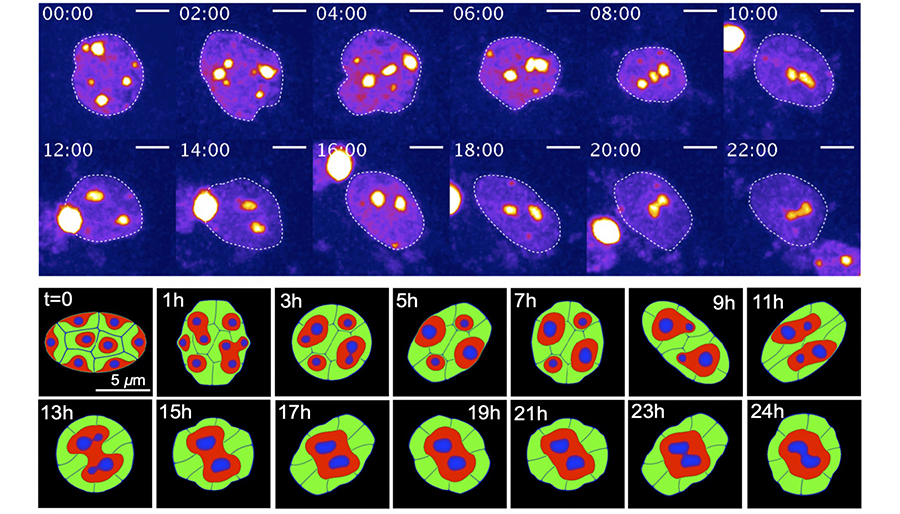
Seirin-Lee, S., Osakada, F., Takeda, J., Tashiro, S., Kobayashi, R., Yamamoto, T., & Ochiai, H. (2019). Role of dynamic nuclear deformation on genomic architecture reorganization. PLOS Computational Biology, 15(8), e1007289. https://doi.org/10.1371/journal.pcbi.1007289
Kuwamura, M., Seirin-Lee, S., & Ei, S.-I. (2018). Dynamics of localized unimodal patterns in reaction-diffusion systems related to cell polarization by extracellular signaling. SIAM Journal on Applied Mathematics, 78(6), 3238–3257. https://doi.org/10.1137/18M1163749
Seirin-Lee, S., Tashiro, S., Awazu, A., & Kobayashi, R. (2017). A new application of the phase-field method for understanding the reorganization mechanisms of nuclear architecture. Journal of Mathematical Biology, 74, 333–354. https://doi.org/10.1007/s00285-016-1031-3
Seirin-Lee, S. (2016). Lateral inhibition-induced pattern formation controlled by the size and geometry of the cell. Journal of Theoretical Biology, 404, 51–65. https://doi.org/10.1016/j.jtbi.2016.05.025


