研究概要


1塩基レベルの機能ゲノミクス
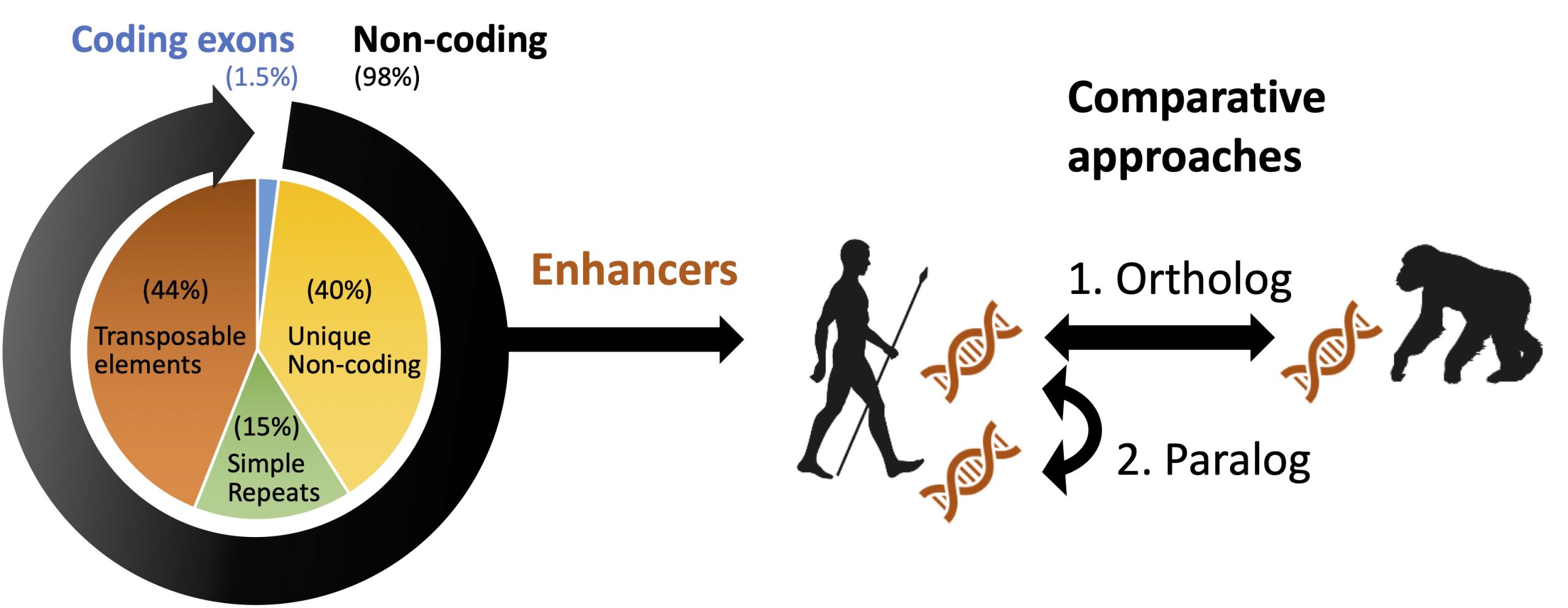
ヒトゲノムの 98%はノンコーディング領域によって占められており、その機能の大部分は依然として不明です。近年の様々な研究から、近傍遺伝子の転写を制御する「エンハンサー」がノンコーディング領域内に多数存在し、またその変異や多型が遺伝性疾患や進化の主要因となりうることが分かってきています。すなわち、疾患や進化そしてゲノムの本質的理解に近づくには、ノンコーディングゲノムに隠されたエンハンサーを解析し、遺伝子制御機構の全体像(レギュローム)を明らかにする必要があります。
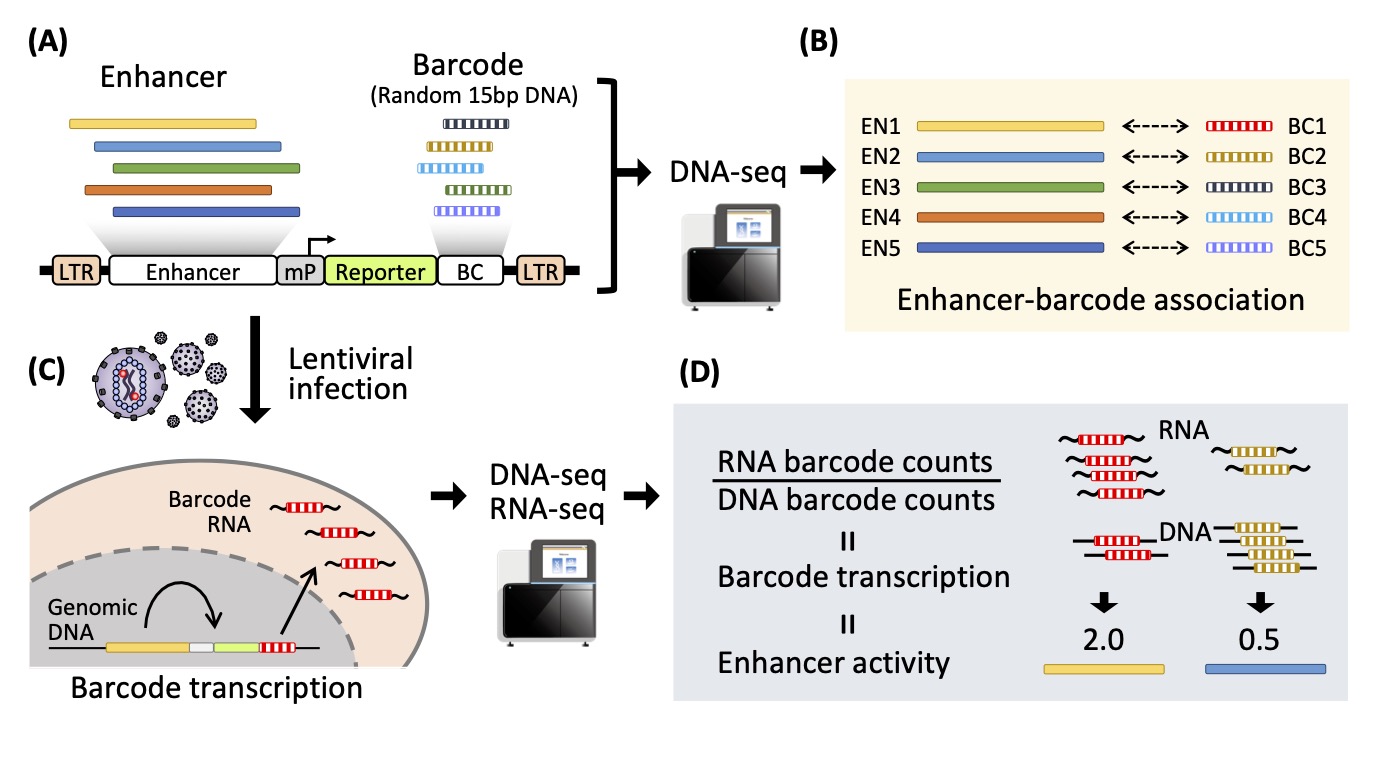
井上がこれまで開発してきた大規模並列レポーターアッセイ(lentiMPRA)は、転写バーコードを用いることにより、十数万のエンハンサーバリアントの活性を一度の実験で大規模並列的かつ定量的に解析できる画期的技術であり、第4期ENCODEプロジェクトに活用されるなど高く評価されています。私達は、lentiMPRAやCRISPR activation/interferenceといった新しい機能ゲノミクス技術を駆使することで、進化や疾患に関わるエンハンサーの機能を高解像度で解析し、ヒトレギュロームの本質的理解を目指しています。
種間 / 種内比較機能ゲノミクス
我々ヒトをヒトたらしめている遺伝的要因は何なのか?私達は、ヒト系譜において独特に進化してきた機能ゲノム領域を発見するために、2方向の比較ゲノミクスアプローチをとっています。
1. オーソログ比較
霊長類種間(ヒト、チンパンジー、カニクイザルなど)のゲノム・エピゲノム比較により、種間で異なるエンハンサー領域を同定・解析することで、ヒト特性とヒトゲノム進化を理解します。
2. パラログ比較
ヒトゲノム内に存在する転移因子に注目します。転移因子は真核生物の進化の過程でゲノムに組み込まれた後に増幅し、ヒトゲノムのおよそ半分を占めるに至っています。また多くの転移因子ファミリーがエンハンサー機能を持つことが示唆されており、パラログエンハンサー解析の格好のモデルと言えます。
このような目的のために、私達はRNA-seq, ATAC-seq, Cut&Tag, Nanopore long-read DNA-seqといった様々なゲノム・エピゲノム解析技術を駆使しています。